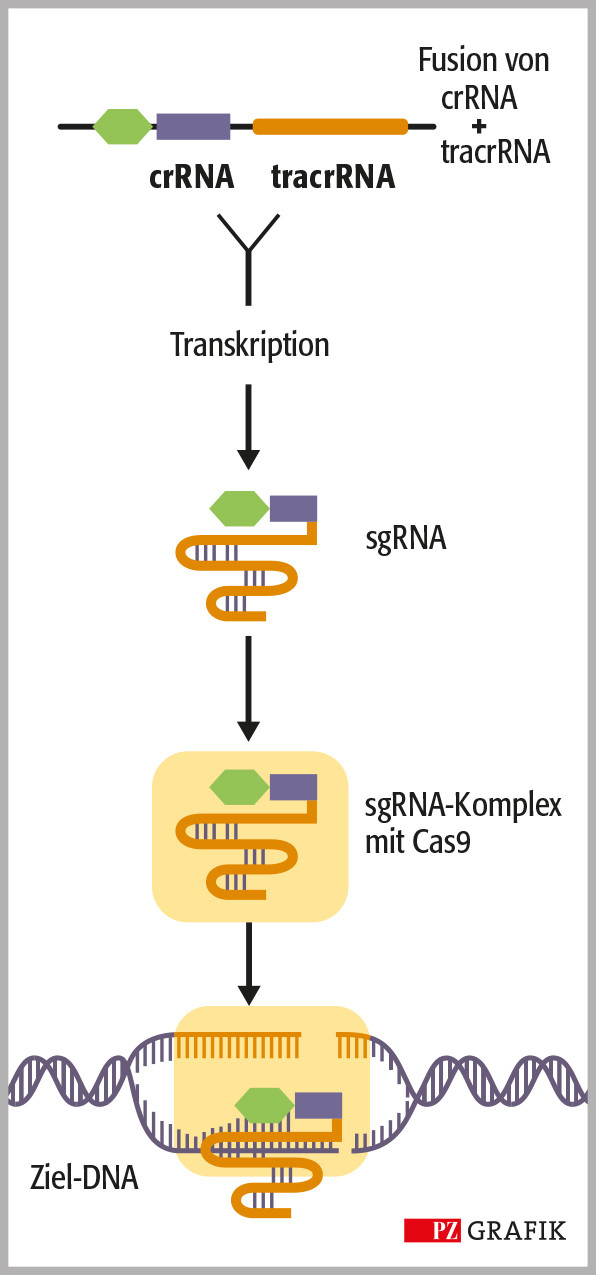
In einer berühmt gewordenen Publikation in dem Wissenschaftsmagazin »Science« mit dem Titel »A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity« aus dem Jahr 2012, beschreiben Doudna und Charpentier zusammen mit Kollegen nicht nur das CRISPR/Cas-System als solches, sondern auch eine bahnbrechende Vereinfachung des Systems. Sie zeigten, dass die crRNA und die tracrRNA zu einzelnen Leit-RNA (sgRNA) fusioniert werden können (Abbildung 1B), was die CRISPR-Technologie massiv vereinfachte und zu einer Art Universalwerkzeug zum Umschreiben der Erbsubstanz von Bakterien, Pflanzen, Tieren und Menschen machte.
Diese Arbeit markiert den Beginn der CRISPR-Ära und war maßgeblich dafür verantwortlich, dass die beiden Forscherinnen 2020 mit dem Nobelpreis für Chemie ausgezeichnet wurden. Zu Recht wohlgemerkt, denn letztlich ist es ihrer Forschung zu verdanken, dass Wissenschaftler heute in der Lage sind, auf einzigartige Weise in den Bauplan des Lebens einzugreifen und so gezielt das Erbgut umzuschreiben.
Neben dem Cas9-System werden inzwischen auch Cas12, Cas14 und Cas13 häufiger genutzt. Sowohl Cas12 als auch Cas14 sind (wie Cas9) RNA-gelenkte DNasen, die neben doppelsträngiger DNA (dsDNA) auch Einzelstrang-DNA (ssDNA) spalten. Cas12 hat eine höhere Spezifität für dsDNA, während Cas14 eine höhere Spezifität für ssDNA besitzt. Cas13 ist dagegen eine RNA-gelenkte RNase, die einzelstängige RNA (ssRNA) spaltet.