Messenger RNA (mRNA):
Mit den Impfstoffen Spikevax® und Comirnaty® gegen COVID-19 erhielten die ersten mRNA-Arzneistoffe die Zulassung. Der Ansatz ist, dass eine bestimmte mRNA in Zellen eingebracht und dort translatiert wird. Dadurch entsteht vorübergehend (transient) das entsprechende Protein, das beispielsweise als Antigen zur Stimulation des Immunsystems dient. Für eine Protein-Ersatztherapie, beispielsweise bei Hämophilie A, ist die Methode eher ungeeignet, weil es zu erheblichen Nebenwirkungen durch Off-target-Effekte kommen kann.
Antisense-Oligonukleotide (ASO):
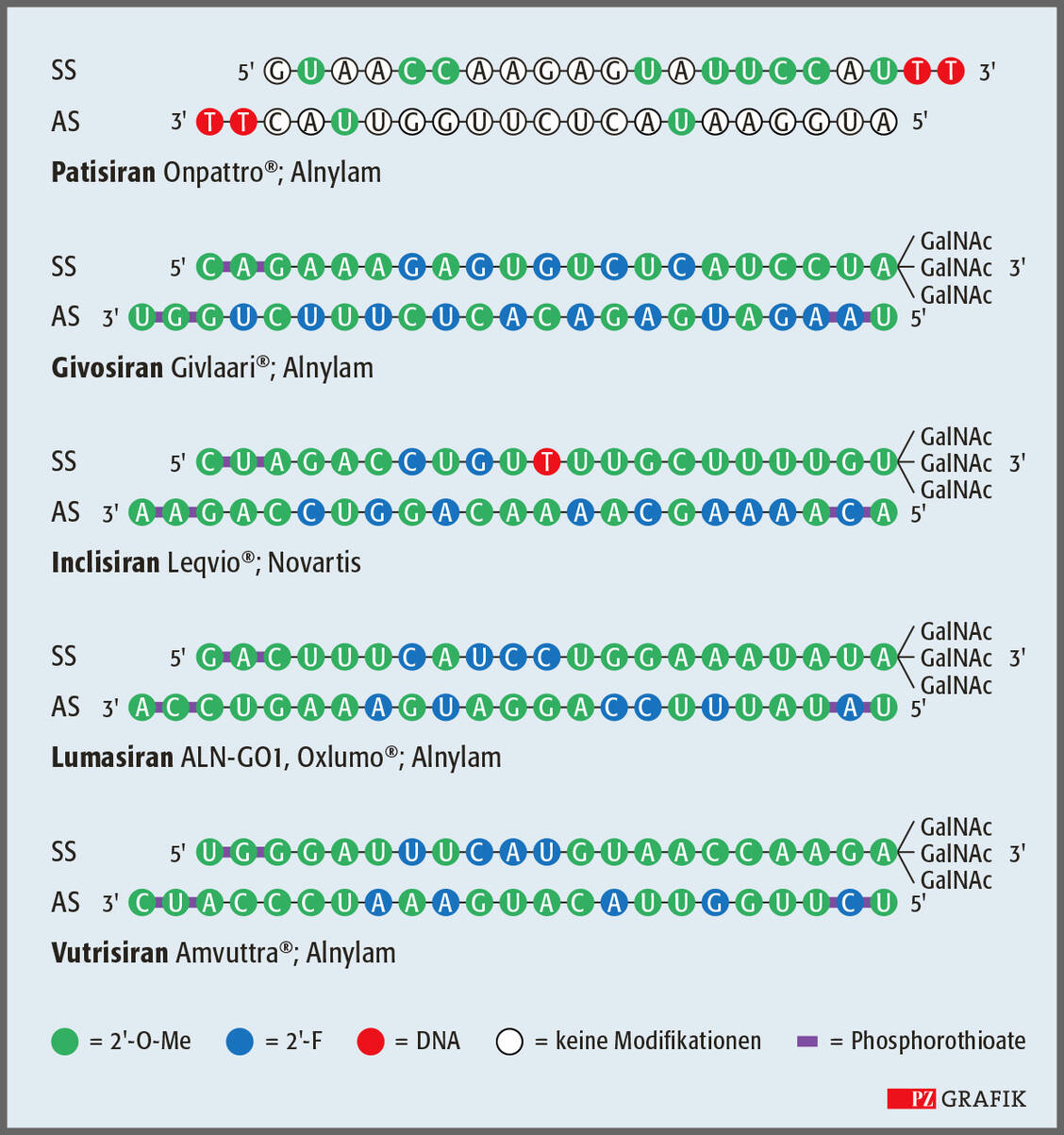
Einzelsträngige RNA- oder DNA-Oligonukleotide werden in die Zelle eingebracht und binden dort an die zu ihrer eigenen Sequenz komplementäre mRNA (»Sense«-Molekül). Durch diesen doppelsträngigen Bereich kommt es zu einer sterischen Blockade der Funktionalität. Je nachdem, welche Sequenz für das Antisense-Oligonukleotid gewählt wurde und in welcher Region der mRNA die Bindung stattfindet, kommt es zur Inhibition des Translationsstarts, zur Beeinträchtigung des mRNA-Spleißprozesses (Beispiel: Nusinersen in Spinraza®) oder zum Abbau der Ziel-mRNA im DNA-RNA-Komplex durch RNase H (Beispiel: Inotersen in Tegsedi®, Volanesorsen in Waylivra®).
Micro RNA (miRNA):
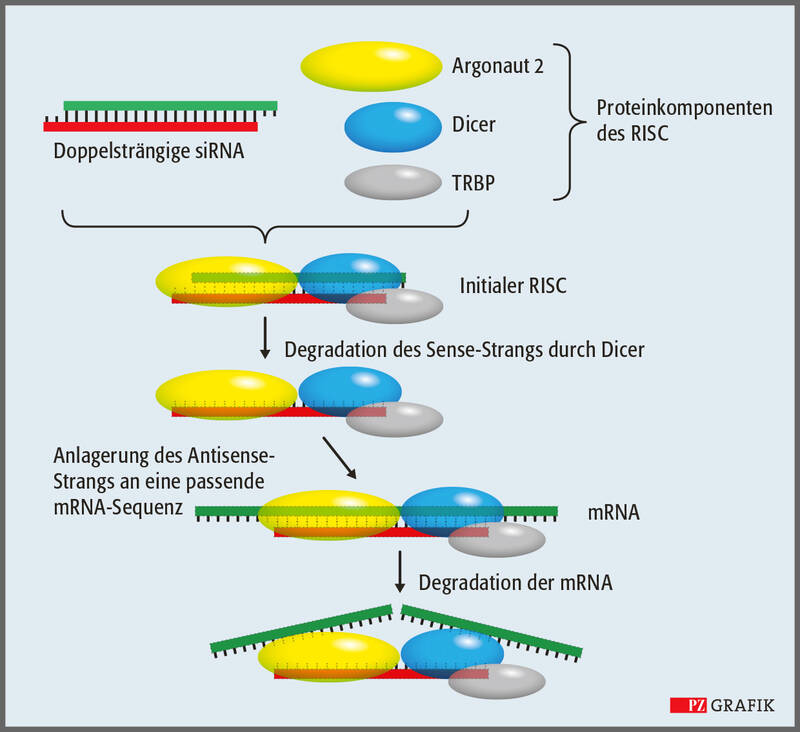
Kurze, 20 bis 25 Nukleotide lange, doppelsträngige RNA, die natürlicherweise in Zellen vorkommt und dort die Translation bestimmter mRNA steuert. Therapeutisch eingesetzt imitieren die miRNA häufig ihre natürlichen Pendants und stellen deren Funktionalität, beispielsweise in Tumor-zellen, wieder her. Die Wirkweise ähnelt der von siRNA (siehe Haupttext) und führt zur Zerstörung der adressierten mRNA. Im Gegensatz zu siRNA binden miRNA bereits bei einer Teilkomplementarität an die mRNA, vor allem im Bereich der 3'-UTR (nicht translatierte Region). Dadurch kann eine miRNA mehrere unterschiedliche mRNA hemmen.
RNA-Aptamere:
Kurze einzelsträngige RNA-Moleküle, die weniger aufgrund ihrer Sequenz als vielmehr wegen ihrer dreidimensionalen Struktur an unterschiedliche Zielmoleküle binden, zum Beispiel Kohlenhydrate, Proteine oder Nukleinsäuren. Aptamere können dort entweder als Agonisten, Antagonisten oder allgemeine Bindepartner wirken. Das inzwischen wieder vom Markt genommene Pegaptanib (Macugen®) war gegen VEGF gerichtet und zur Therapie der altersabhängigen feuchten Makuladegeneration zugelassen.
Short activating RNA (saRNA):
Kurze, 21 Nukleotide lange, doppelsträngige RNA, von der durch das Protein AGO2 (Argonaut 2) ein Strang (Passenger-Strang) geschnitten wird. Der verbleibende Strang bindet im Komplex mit AGO2 an bestimmte Promotoren von Genen und verstärkt deren Transkription.
Single guide RNA (sgRNA):
Mit dem CRISPR-Cas-System steht ein recht effizientes Werkzeug zur Genomveränderung zur Verfügung. Die Cas-Nuklease benötigt für das zielgenaue Schneiden der DNA in einer Zelle eine sgRNA, die sie an ihre komplementäre Sequenz führt.