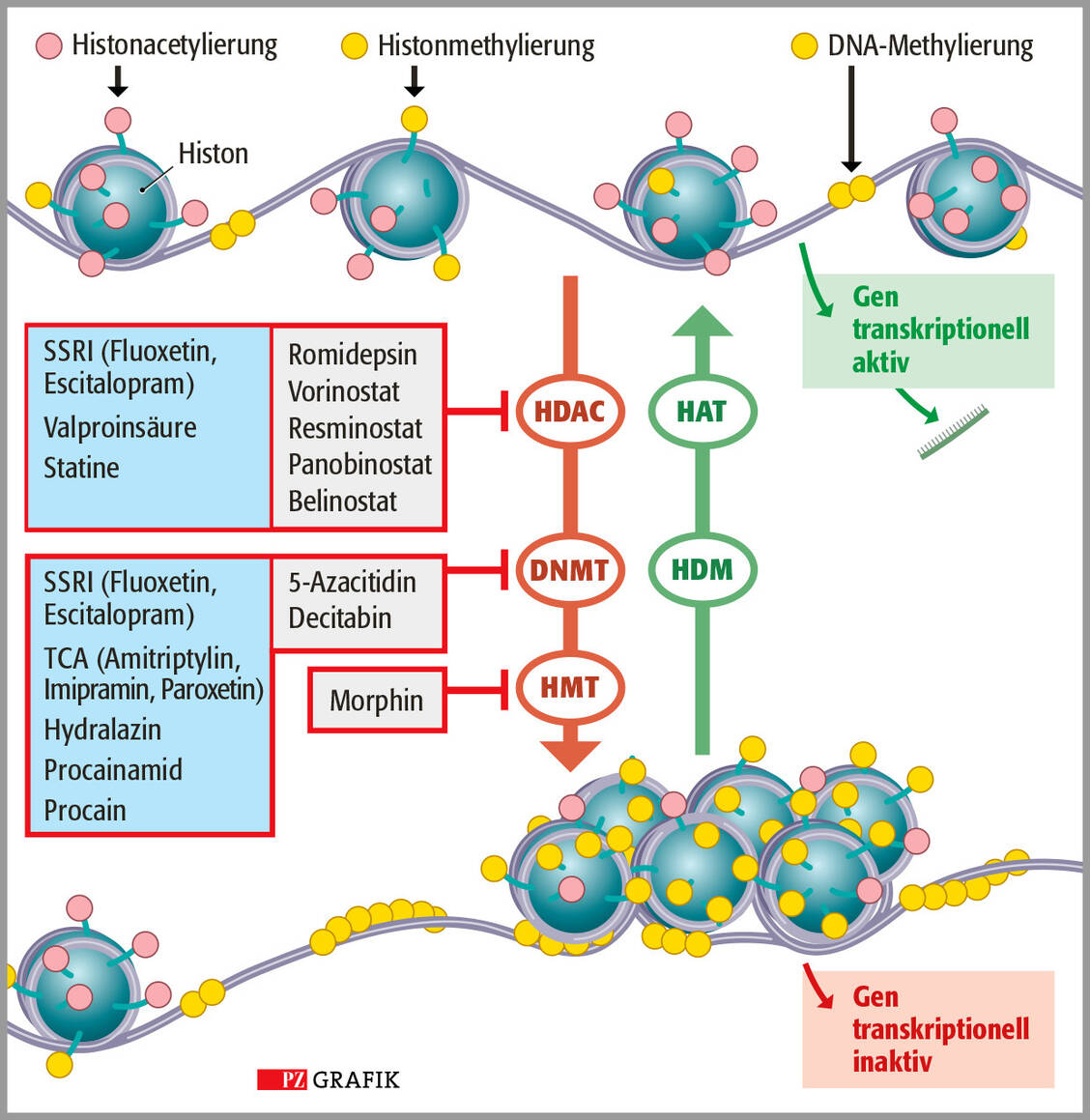
Auch die im Darm lebenden Mikroorganismen beeinflussen über ihre Metabolite das Epigenom. Hier stehen insbesondere kurzkettige Fettsäuren (short-chain fatty acids, SCFA) wie Acetat, Butyrat und Propionat im Fokus, die im Darm durch Fermentation von Ballaststoffen gebildet werden und auf Histonacetylierung und -methylierung einwirken. Untersuchungen im Mausmodell beschreiben unterschiedliche Effekte je nach ballaststoffreicher (polysaccharidreicher) Ernährung und westlicher, polysaccharidarmer Ernährung (20).
Der Zusammenhang von Mikrobiom und Immunsystem wird ebenfalls umfassend untersucht und die Aktivierung der Immunzellen mit epigenetischen Signaturen in Verbindung gebracht. Monozyten, Makrophagen und NK-Zellen des angeborenen Immunsystems können eine Art Training durchlaufen, das zu einer verbesserten ersten schnellen Immunantwort bei Infektionen führt. Dieses Training kann beispielsweise durch ß-Glucan angetrieben werden, ein Polysaccharid, das natürlicherweise in den Zellwänden von Getreide, Hefen, Bakterien und Pilzen vorkommt. In vitro zeigten sich in verschiedenen Genclustern der Immunzellen verstärkt Histonmodifikationen (21). Inwieweit sogenannte Immunstimulanzien wie Echinacea- und Zistrosenextrakte, Vitamin C, Zink, Probiotika, verschiedene Substanzen der Phytotherapie oder Stress epigenetische Mechanismen beeinflussen, ist in der wissenschaftlichen Literatur noch umstritten (14, 15, 16).