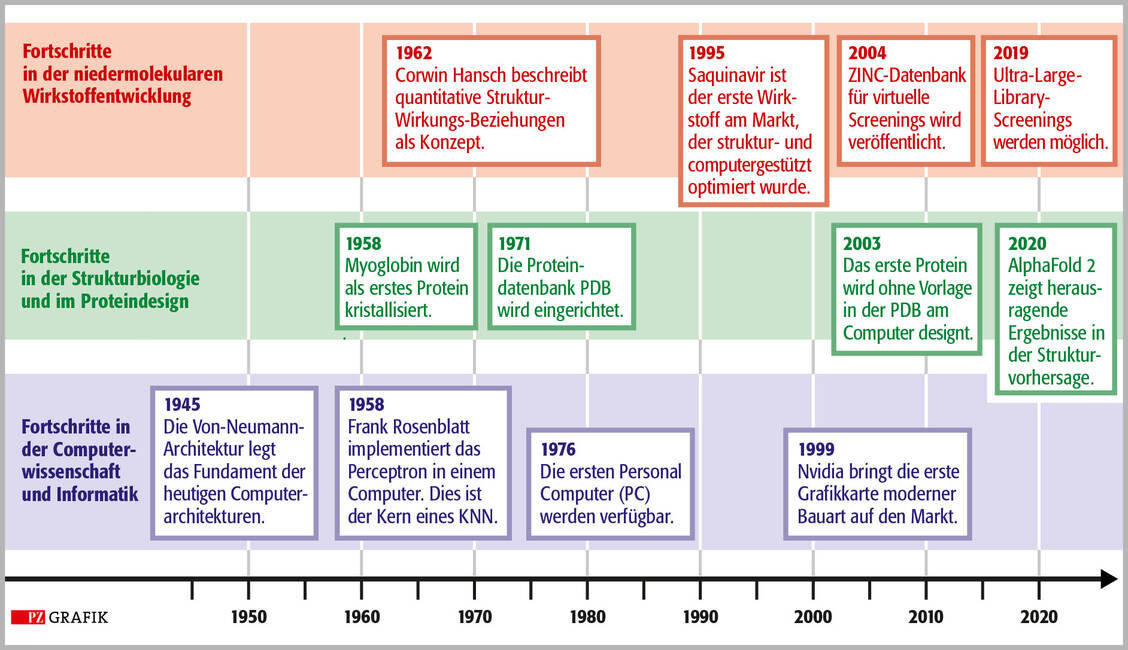
Künstliche Intelligenz umfasst per Definition der Bundesregierung Methoden zur Lösung von Anwendungsproblemen in der Mathematik und Informatik, die zur Selbstoptimierung fähig sind. Dazu zählen unter anderem Methoden des maschinellen Lernens, aber auch wissensbasierte Systeme.
Maschinelles Lernen bezeichnet die Verwendung von computergestützten Methoden und statistischen Modellen, die Gesetzmäßigkeiten, Abhängigkeiten und Muster aus zugrunde liegenden Daten ableiten. Beispiele für maschinelles Lernen sind künstliche neuronale Netzwerke, Support-Vektor-Maschinen sowie die Anwendung von linearer Regression.
Künstliche oder artifizielle neuronale Netzwerke (KNN oder ANN) wurden nach dem Beispiel des Neurons im Nervensystem entwickelt. Anhand von Testdaten werden Gewichtungen von verschiedenen Parametern bestimmt, wobei das Ergebnis der Zuordnungsvorhersage dazu dient, die Gewichte so lange anzupassen, bis die Zuordnung optimal ist. Ähnlich zu der Verschaltung von Neuronen im menschlichen Gehirn können diese Netzwerke verschiedene nacheinander geschaltete Neuronen – und damit mehrere Schichten – haben.
Deep Learning oder mehrschichtiges Lernen ist eine Unterordnung der maschinellen Lernverfahren. Sie basieren auf KNN, die viele Schichten haben. Dadurch können deutlich mehr Parameter verarbeitet und berechnet werden, es werden aber auch große Datensätze benötigt. Ein Nachteil ist, dass die Optimierung in den Schichten bisher schwer oder gar nicht nachzuvollziehen ist.